В субботу 11 октября питонист Александр Ильин прочитал великолепный вебинар по анализу данных на Python. Мы решили собрать полезные и эффективные методы для обработки таблиц в Pandas и поделиться ими с вами. Теперь не придется гуглить или спрашивать у чата гопоты, как сделать то-сё, пятое, десятое. Забирайте и используйте в своей работе, доводя навыки до автоматизма.
🐸 Как начать вкатываться в питон? Благоприятная среда
Прежде чем начать, напомним, что самая сложная часть для новичка — это установка нужного программного обеспечения. Рекомендуемые инструменты от Александра Ильина:
- Anaconda: дистрибутив, содержащий Python и сразу большой набор библиотек (Pandas, библиотеки для рисования).
- Jupyter Notebook: среда разработки, хорошо подходящая для аналитических задач, рисования графиков и интерактивной работы с кодом.
- Google Colab: альтернатива юпитеру, не требующая установки, работает онлайн на серверах Google, но для работы с вашими файлами потребуется выгрузить их на Google Drive и прописать путь к ним.
- PyCharm / VS-Code: более навороченные среды для написания кода — не рекомендуются как первые для новичков.
🐍 Python vs Excel
Python — один из самых распространенных языков программирования, который завоевал популярность благодаря относительной простоте по сравнению с другими языками и большому комьюнити, что обеспечивает много готовых решений для различных задач, включая веб-разработку, машинное обучение и анализ данных. Последнее — то, чем мы сегодня и займемся.
🐼 Работа с табличками в Pandas
На вебинаре мы анализировали данные о встречаемости рачков (копепод) в разных местах, на разных глубинах и в разных хозяевах. Изначально данные представлены в виде набора 7 однотипных excel-таблиц с большим количеством строк и 189 колонками. Давайте повторим действия и запомним полезные фишки.
Ссылки на все датасеты:
- www.gbif.org/dataset/21afc06a-f097-47bd-b85e-ff2301436101
- www.gbif.org/dataset/6254b79a-5df5-4ae1-b1f0-2c8e8969b73e
- www.gbif.org/occurrence/download/0015916-250711103210423
- www.gbif.org/dataset/34135c23-a595-43bc-b3f0-a08a4748d861
- www.gbif.org/dataset/0d0f1fc9-3dff-435d-ad51-907485ea584c
- www.gbif.org/dataset/dffe30bd-5871-4044-b5fe-d20fee674573
- www.gbif.org/dataset/be8f0b51-2030-4402-80e9-01095875de64
🧹 Очистка и преобразование данных
Сперва мы подгрузим необходимые библиотеки:
import pandas as pd
from pathlib import Path
import seaborn as sns
import matplotlib.pyplot as plt📥 Загрузка и объединение таблиц. С помощью кода можно считать таблицы из папки и объединить в один датафрейм, независимо от их количества (хоть 7, хоть 700). Вот как мы делаем c excel-файлами:
# Указываем путь к папке, где лежат Excel-файлы
path = 'data/gbif/'
# Преобразуем строку пути в объект Path (из библиотеки pathlib)
path = Path(path)
# Создаем пустой список, куда будем сохранять считанные датафреймы
dfs = []
# Перебираем все файлы с расширением .xlsx в указанной папке
for gbif in path.glob('*.xlsx'):
# Считываем из каждого Excel-файла лист с названием 'GBIF' в датафрейм
df = pd.read_excel(gbif, sheet_name='GBIF')
# Добавляем считанный датафрейм в список dfs
dfs.append(df)
# Объединяем все датафреймы из списка dfs в один общий датафрейм
# pd.concat "склеивает" их по строкам (по умолчанию axis=0)
df = pd.concat(dfs)Библиотеки os и pathlib мы подробно обсуждаем на курсе по питону на занятии «Биологические объекты и файловые пути»
Если у вас файлы другого формата, например, csv, то в Pandas есть соответствующие функции:
# Загружает CSV-файл
# Аргумент index_col указывает, какой столбец использовать в качестве индекса
df = pd.read_csv('file.csv', index_col='index_column')
# Загружает TSV-файл (таблица, где значения разделены табуляцией '\t')
# Аргумент sep='\t' задает разделитель столбцов
df = pd.read_csv('file.tsv', sep='\t')
# Загружает JSON-файл
# Pandas автоматически преобразует данные JSON в табличную структуру, если это возможно
df = pd.read_json('file.json')df — это типичное имя переменной для DataFrame. Датафрейм — основная двумерная структура данных библиотеки Pandas, напоминающая таблицу в Excel или SQL. Rаждый столбец в DataFrame — отдельный объект типа Series.
🔍 Просмотр данных. Мы считали данные, смотрим на них. Вот самые удобные методы:
# По умолчанию показывает первые пять строк датафрейма
df.head()
# По умолчанию показывает последние пять строк датафрейма
df.tail()
# Возвращает одну случайную строку из датафрейма
df.sample()
# Выводит базовую статистику по числовым столбцам:
# количество значений (count), среднее (mean), стандартное отклонение (std),
# минимумы (min), максимумы (max), квартили
df.describe()
# Отображает краткую сводку о датафрейме:
# количество строк и столбцов, названия колонок, типы данных,
# количество ненулевых (ненулевых/ненановых) значений и общий объем памяти
df.info()🧾 Отбор колонок. Для упрощения работы отбираем только необходимые колонки из почти 200.
# Атрибут, который показывает список всех колонок в датафрейме
df.columns
# Создаем список с названиями нужных колонок
# Многострочная строка ('''...''') тут очень удобна
cols = '''
occurrenceID
catalogNumber
...
eventDate
associatedOccurrences
associatedTaxa
'''.splitlines()
# Убираем возможные лишние пробелы и пустые строки из списка
cols = [c.strip() for c in cols if c]
# Оставляем в датафрейме только указанные колонки
df = df[cols]💩 Данные в реальной жизни — грязные. И наиболее распространенная причина ошибок — это человеческий фактор. Нам же важно получить гомогенные колонки, поэтому займемся очисткой данных.

❌ Удаление дубликатов. Распространенная проблема — дубликаты. Например, вы объединяли таблицы и у вас сохранились одинаковые записи. Попробуем удалить дубликаты с помощью одноименного метода. Сперва отберем колонки интереса:
search_duplicates = '''scientificName
associatedTaxa
eventDate
decimalLatitude
decimalLongitude
associatedReferences'''.split()Посмотрим, сколько строк было в таблице:
df.shape[0]
# 14091Дропнем дубликаты:
df = df.drop_duplicates(subset=search_duplicates)И проверим, как изменилась форма датафрейма:
df.shape[0]
# 10244Было удалено почти 4000 строк!
🔪 Обработка пропусков. Отдельный челлендж — это пропуски (NaN). Самый простой способ — удалить строки с пропусками, если их мало. Код ниже убирает все строки из колонки 'minimumDepthInMeters', где были пропущенные значения:
df.minimumDepthInMeters.dropna()Другие методы включают заполнение NaN средними/медианами по соответствующим подгруппам (imputation) или использование моделей машинного обучения для предсказания пропущенных значений. В некоторых случаях пропуск может означать «ниже порога определения», и тогда его заменяют нулем df.fillna(0)
🔧 Конвертировать тип колонки. В колонке, которая кажется числовой, могли встречаться дробные числа, записанные не по стандартам питона. В питоне разделитель между целой и дробной частью — точка (например, 0.5). Но в колонке невнимательные товарищи могли записать через запятую (0,5), тогда такая колонка будет воспринята при считывании файла как object. Как это пофиксить? Заменим в колонке все запятые на точки:
df.minimumDepthInMeters = df.minimumDepthInMeters.str.replace(',','.')В колонке могут еще быть записаны единицы измерений, например, '30 см'. Дабы избавиться от них мы разделим значения в колонке по пробелу и отберем по индексу первое значение:
df.minimumDepthInMeters = df.minimumDepthInMeters.str.split().str[0]Еще проблема: записанные значения через дефис '1.5-2'. Обработаем аналогично предыдущему, только разделим по дефису '-':
df.minimumDepthInMeters = df.minimumDepthInMeters.str.split('-').str[0]Вот теперь можно перевести колонку в формат числа с плавающей точкой. Метод astype() позволяет изменить тип данных элементов в массиве или серии:
df.minimumDepthInMeters = df.minimumDepthInMeters.astype(float) Далее можно проводить статистический анализ в колонке, например, посчитать среднее:
df.minimumDepthInMeters.mean()
# 13.502396271008395🎨 Рисуем графики
Пришло время визуализировать наши данные. Если вы работали в Excel, то наверняка знаете, что можно построить график, выделив определенные диапазоны данных и нажав несколько кнопок, где мы указываем, что должно идти по оси X, что по оси Y, какой тип графика, цветовую схему.
В Python — всё иначе, что поначалу может показаться непривычным. Специальными командами мы описываем, что хотим. Это формализированный язык: в нем строгие правила и кажется, что это дольше, чем сделать руками в Excel. На самом деле в перспективе код экономит время, потому что единожды написав его для получения графика, вам больше не нужно будет создавать его заново. Вы можете подставлять разные данные такого же формата, вызывать код, и вуаля, картинки будут сами перестраиваться!
В Python — всё иначе, что поначалу может показаться непривычным. Специальными командами мы описываем, что хотим. Это формализированный язык: в нем строгие правила и кажется, что это дольше, чем сделать руками в Excel. На самом деле в перспективе код экономит время, потому что единожды написав его для получения графика, вам больше не нужно будет создавать его заново. Вы можете подставлять разные данные такого же формата, вызывать код, и вуаля, картинки будут сами перестраиваться!
📊 Гистограмма распределения глубин. Сперва построим график, как часто организм встречается на определенной глубине:
# Строим гистограмму распределения значений из столбца 'minimumDepthInMeters'
# Аргумент bins=20 задает количество корзинок: чем больше значение, тем детальнее гистограмма
sns.histplot(df.minimumDepthInMeters, bins=20)
# Добавляем сетку на фон графика для лучшего восприятия
plt.grid()
# Подписываем ось X как 'Depth'
plt.xlabel('Depth')
# Подписываем ось Y как 'Number of occurrences'
plt.ylabel('Number of occurrences')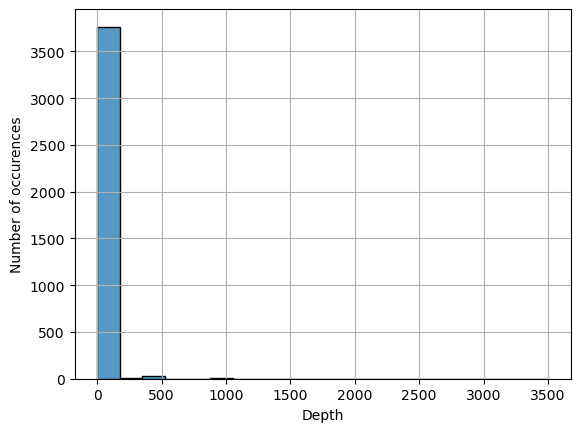
Большинство объектов найдено на глубине 0–100 м. Но смотреть на график неудобно, потому что большие значения количества наблюдений забивают весь масштаб.
⚙️ Кастомизация графиков. Когда на графике есть очень большое и очень маленькое значения, можно применить логарифмическую шкалу — в данном случае для оси Y, что позволяет лучше увидеть маленькие значения рядом с большими (например, единичные находки на 3500 м). И дальше оперировать порядками величин.
sns.histplot(df.minimumDepthInMeters, bins=20)
plt.grid()
# Меняем масштаб оси Y на логарифмический
plt.yscale('log')
plt.xlabel('Depth')
plt.ylabel('Number of occurrences')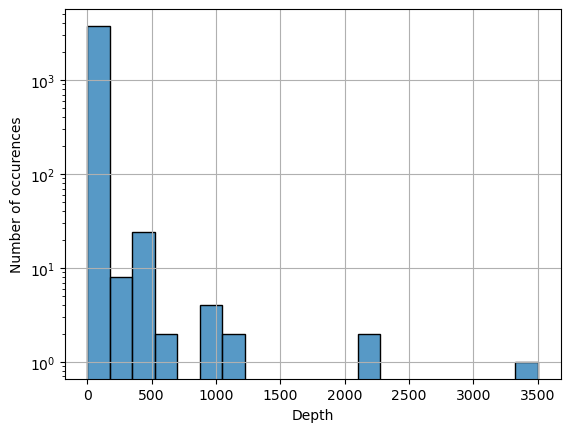
🧠 Более сложное преобразование таблицы
💫 Связь копепод и их хозяев. Чтобы нарисовать еще более продвинутые графики, нам понадобится преобразовать исходную таблицу. Наша таблица — длинная, в ней в одних строках записаны рачки, в других — их хозяева. Нам нужно соотнести рачков и их хозяев, чтобы произвести подсчет числа уникальных сочетаний. Мы будем менять форму таблицы, перенеся часть информации в строках о хозяевах в строки с симбионтами. Примерно так, как нарисовал Александр:
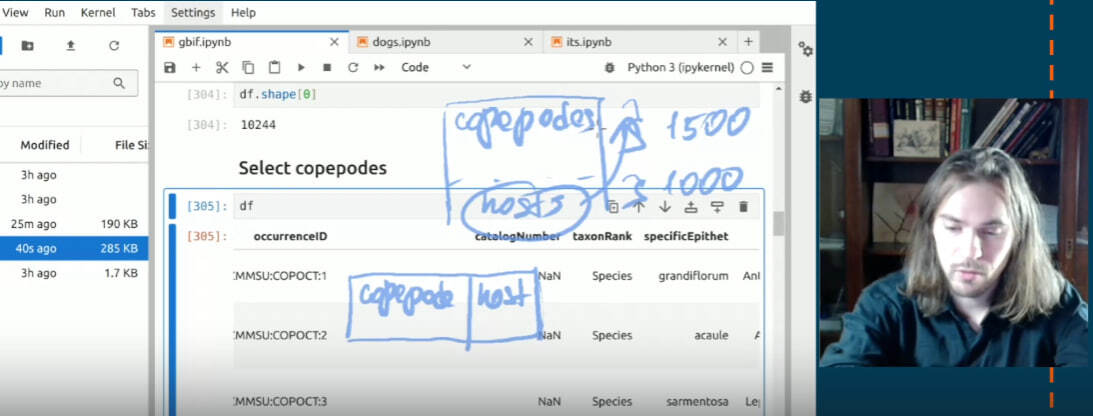
В таблице есть колонка 'class' по которой мы можем понять перед нами рачок или хозяин.
# Получаем столбец 'class' — он содержит класс животных
df['class']
# 0 Anthozoa
# 1 Anthozoa
# 2 Anthozoa
# 3 Anthozoa
# 4 Anthozoa
# ...
# 3091 Copepoda
# 3101 Copepoda
# 3102 Copepoda
# 3103 Copepoda
# 3222 Copepoda
# Name: class, Length: 10244, dtype: objectМы создаем новую булевую колонку, в которой у рачков (называются 'Hexanauplia') будет True, а их хозяев — False.
# Добавляем новый столбец 'is_copepode', который отмечает строки, где класс равен 'Hexanauplia'.
# Операция df['class'] == 'Hexanauplia' возвращает серию True/False.
# Эти булевы значения записываются в новый столбец, создавая логический признак
df['is_copepode'] = df['class'] == 'Hexanauplia'Посмотрим, сколько вообще рачков получилось:
df.query('is_copepode == True').shape
# (4296, 35)Совмеситим рачков с их хозяевами с помощью ID-шников (должны совпадать идентификаторы 'occurrenceID' у хозяев и 'associatedOccurrences' у копепод).
# Вывести содержимое колонки 'associatedOccurrences'
print(df['associatedOccurrences'])
# 0 NaN
# 1 NaN
# 2 NaN
# 3 NaN
# 4 NaN
# ...
# 3091 urn:catalog:ZMMSU:COPHEX:1121
# 3101 urn:catalog:ZMMSU:COPHEX:1140
# 3102 urn:catalog:ZMMSU:COPHEX:1145
# 3103 urn:catalog:ZMMSU:COPHEX:1146
# 3222 urn:catalog:ZMMSU:COPHEX:914
# Name: associatedOccurrences, Length: 10244, dtype: object
Здесь Александр использовал продвинутую конструкцию (method chaining), где df обернут в скобки, чтобы последовательно применять разные методы, записывая их с новой строки. Это напоминает %>% в dplyr.
# Отбираем только те строки, где is_copepode == True
df = (
df.query('is_copepode == True')
# Объединяем датафрейм сам с собой, чтобы "сопоставить" копепод с их хозяевами
.merge(
# Оставляем только нужные столбцы для соединения:
# occurrenceID — уникальный идентификатор,
# associatedOccurrences — ссылка на связанные записи,
# class — таксономический класс
df[['occurrenceID', 'associatedOccurrences', 'class']],
# Соединяем таблицы:
left_on='occurrenceID',
right_on='associatedOccurrences',
# Добавляем суффикс "_host" к столбцам из правой таблицы,
# чтобы отличать их от исходных
suffixes=['', '_host']
)
)Теперь подсчитаем количество уникальных пар рак-хозяин по таксономическому уровню:
# Группируем датафрейм по комбинации значений в столбцах 'order' и 'class_host'
# .size() считает количество строк (наблюдений) в каждой группе
pair_counts = df.groupby(['order', 'class_host']).size().reset_index(name='count')
# Просматриваем результирующий датафрейм
pair_counts
# order class_host count
# 0 Canuelloida Malacostraca 5
# 1 Cyclopoida Anthozoa 87
# 2 Cyclopoida Ascidiacea 2
# 3 Cyclopoida Asteroidea 487
# 4 Cyclopoida Bivalvia 28
# 5 Cyclopoida Cephalopoda 1
# ...
# 27 Siphonostomatoida Malacostraca 2
# 28 Siphonostomatoida Ophiuroidea 147🧮 Скаттерплот. Дальше построим скаттерплот, где размер точки пропорционален количеству найденных ассоциаций между конкретным классом хозяина и отрядом рака:
# Строим диаграмму рассеяния
sns.scatterplot(
data=pair_counts, # Источник данных — датафрейм pair_counts
x='class_host', # Ось X — таксономический класс хозяина
y='order', # Ось Y — отряд копепод (Copepoda)
size='count' # Размер точек соответствует количеству наблюдений (count)
)
# Добавляем сетку для лучшего восприятия структуры графика
plt.grid()
# Поворачиваем подписи оси X на 90°, чтобы они не перекрывались
plt.xticks(rotation=90)
# Подписываем оси для ясности
plt.xlabel('Host class')
plt.ylabel('Copepoda order')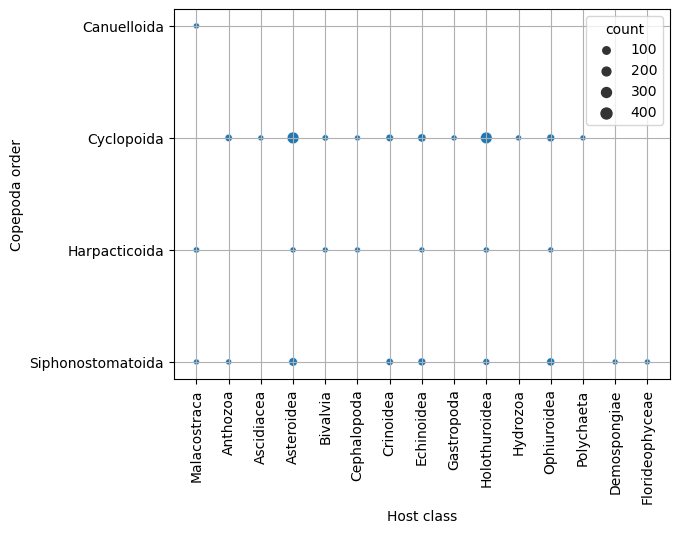
Когда мы строим график, это нормально, что вначале он получается не таким, каким мы хотим его видеть. Это вполне обыденная вещь. Если писать только минимальную команду sns.scatterplot(), то по дефолту график будет без решетки, которая удобна для восприятия и сразу показывает, к каким значениям относятся, например, границы. Также подписи к классам насладывались бы друг на друга, если бы их не повернули.
Строя график, мы постепенно кастомизируем его, добавляя код. Чем больше вы добавите кода, тем более навороченный будет ваш график. Иногда это необходимо, если вы делаете финальные презентации в журнал. Иногда это не обязательно, если вы быстро хотите посмотреть, как выглядят ваши данные и показать коллегам.
📦 Boxplot. Настала пора построения ящиков с усами для отображения распределения глубин обитания для разных классов хозяев. Мы предварительно уменьшим наш датафрейм, отобрав только те значения, где больше 5 случаев ассоциации для одного хоста, чтобы было хоть какое-то распределение.
# Считаем количество каждого хозяина в таблице с уникальными ассоциациями
host_counts = df['class_host'].value_counts()
# Берем только хозяев, которые встречаются более 5 раз
hosts = host_counts[host_counts > 5].index
# Фильтруем DataFrame, оставляя только строки, где 'class_host' входит в список популярных хостов
df = df.query('class_host in @hosts')Код для непосредственно построения красивого ящика с усами:
# Построения боксплотов:
sns.boxplot(
data=df,
x='class_host',
y='minimumDepthInMeters',
)
# Код для украшения:
plt.grid()
plt.xticks(rotation=90);
plt.xlabel('Host class')
plt.ylabel('Depth')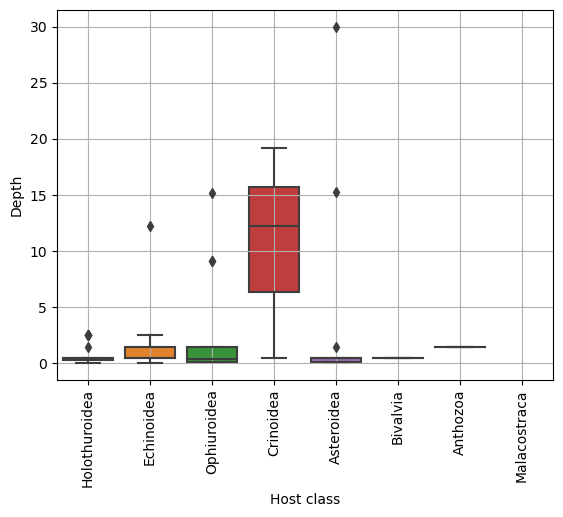
Все вышеперечисленные графики можно было построить с помощью библиотеки matplotlib. Но мы использовали библиотеку Seaborn (фан-факт читать тут), которая представляет собой надстройку над matplotlib с более красивым стилем и дополнительными возможностями. И у нее работает сайт, на котором представлена галерея графиков.
Некоторые графики, а именно гистограммы, теплокарты, барплоты, — это плоть и кровь анализа данных. К этим графикам люди привыкли больше всего, и они очень обширно встречаются. Некоторые графики более экзотичны, они свойственны либо определенной области науки, либо просто данные, на которых их строят, более редкие.
На сайте Seaborn к каждому графику указан код, который его генерирует. Вам не обязательно всегда прописывать самостоятельно все команды для отстройки. Это нормально найти готовый код, скопировать его, возможно, немножко поменять и использовать. Это хорошая практика, ведь вы не потратите свое время.
🦞 Итоги
Мы провели небольшой анализ датасета с рачками. Однако далеко не все занимаются биоразнообразием, экологией и смотрят, какие организмы с кем сожительствуют. Но на самом деле неважно, у вас табличка по метаболомике или по клиническим исследованиям с информацией о том, как прошли испытания какого-то препарата на группах испытуемых — плацебо, лекарства, пол, возраст и так далее. Таблица есть таблица, и поэтому принципы работы с ней сохраняются. С питоном перед вами простор возможностей: фильтровать, транспонировать, делать сводные таблицы (pivot_table) и, конечно, рисовать графики.
🎯 Хотите получить больше полезностей от Ильина? Приходите на следующий вебинар 18 октября в 12:00 мск, где мы прицельно и подробно обсудим другие мастхэвные библиотеки в питоне для биологов и биоинформатиков: зарегистрироваться





